Clique aqui para assistir o vídeo da aula online do dia 4 de junho de 2021. E o chat.
Introdução
Neste tutorial, vamos ampliar o nosso conhecimento sobre as ferramentas que funcionam com R para gerar facilmente relatórios reproduzíveis. Como estas ferramentas podem ser acessadas abertamente pelo público (‘open-source’), podemos aproveitar sem custo do mesmo ambiente assim como milhões de outros pesquisadores.
A primeira ferramenta é o Latex, um programa que compila scripts para o formato bem conhecido de PDF. Sua maior vantagem é a profissionalização dos nossos relatórios - PDF é um formato comum para compartilhar relatórios finais porque eles não podem ser fácilmente editados, e - em contraste com HTML - têm páginas distintas.
Para instalar o Latex no seu computador, o jeito mais fácil é rodar o seguinte código em R (existem muitas versões/distribuições; você não precisa repetir a instalação caso já tenha esta ferramenta no seu computador):
install.packages('tinytex')
tinytex::install_tinytex()
Siga as instruções - por exemplo, vão aparecer alguns ‘erros’ mas pode continuar sem problema; eles não são problemáticos. Depois da instalação, feche o seu R e abra de novo.
Um passo adicional para verificar que tudo está bem configurado é acessar em RStudio Tools -> Global Options -> Sweave -> ‘Weave Rnw files using:’ e escolher ‘knitr’.
Compilando Scripts de R Markdown para PDF
A nossa primeira tarefa não exige nenhuma programação adicional. Na mesma forma que compilamos os nossos documentos com ‘Knit para HTML’ ou ‘Knit para Doc’, a instalação de Latex habilita uma nova opção ‘Knit para PDF’.
Abra um script anterior seu de R Markdown (.Rmd) que compila bem para HTML. Em seguida, escolha a opcão ‘Knit para PDF’. Fácil, não? Como aparece o novo documento? Mais profissional, eu espero.
No futuro, você fica no controle: você poderá compilar para qualquer formato que seja o mais apropriado para o seu relatório.
Relatórios escritos em Latex e compilados para PDF
O que acontece quando compilamos o nosso R Markdown para PDF? O R traduz o nosso script de R Markdown para a linguagem de Latex - literalmente ele gera um arquivo com extensão “.tex” - e depois o programa de Latex interpreta e compila este .tex para PDF.
Isso funciona bem, mas nós estamos habituados para preparar os nossos documentos na linguagem de R Markdown, que é bom e muito fácil de aprender, mas limitado. Por exemplo, se quisermos controlar em detalhe a formatação ou a paginação, não é possível em R Markdown. Então existe uma segunda forma de usar Latex: preparando o nosso script diretamente na linguagem de Latex, e pulando a etapa de R Markdown. O custo é a necessidade de aprender a linguagem de Latex. Dado o uso frequente de Latex no mundo acadêmico para a preparação de manuscritos, artigos e slides, oferecemos aqui um guia preliminar.
O nome para a combinação de R + Latex é ‘Knitr’. Há quatro diferenças fundamentais em usar Knitr em vez de R Markdown.
1. O Tipo de Arquivo
Os nossos scripts de R Markdown tem extensão “.Rmd” e um cabeçalho simples. Quando preparamos um script diretamente em Latex, ele precisa uma extensão “.Rnw”. É sempre melhor começar com um modelo, então começa com “File -> New File -> R Sweave” (ignore o significativo de ‘Sweave’, é um nome velho).
O documento abre com três linhas de texto; os mínimos necessários para um documento de Latex:
\documentclass{article} - O tipo de documento que queremos gerar, o mais comum é um article (artigo). Vamos ver a seguir como gerar apresentações.
\begin{document} - um indicador do começo do conteúdo a ser inserido no documento final. Antes disso podemos inserir pacotes e parâmetros. Depois, digitamos diretamente o seu texto na gramática de Latex.
\end{document} - um indicador do fim do documento. Texto o texto depois ele será ignorado.
2. Compilando o Documento PDF
Escreva algo simples (eg. Hello World!) depois de \begin{document}. Já é um script de Latex/Knitr válido. Como criamos o nosso PDF final a partir desse script? Observe que quando abrimos um arquivo “.Rnw” não há a opção ‘Knit’ em Rstudio. O equivalente é “Compile PDF”. Experimente! Pode demorar um pouco para compilar pela primeira vez, mas, no fim, o R deve ter produzido um PDF, o qual estará salvo na pasta do seu projeto.
3. Formatação de Chunks de Código
A importância de Knitr é que os nossos relatórios contêm os produtos da nossa análise de dados, igual como R Markdown. Mas um ponto chato é que os ‘chunks’ que guardam o nosso código de R têm uma definição diferente.
Para inserir um novo chunk, use a opção ‘+ C’ (ao lado de ‘Run’) em Rstudio. Ele aparece um pouco diferente:
<<>>=
x <- 1+1
x
@Mas, em Rmarkdown, isto significa exatamente o mesmo que:
```{r}
x <- 1 + 1
x
```Como em Rmarkdown, podemos especificar os mesmos parâmetros dos chunks entre <<>>.
<<echo=F, warning=F, message=F>>
Finalmente, você se lembra do nosso código in-line? Ele também é um pouco diferente em Latex/Knitr - precisamos inserir o nome do objeto desejado dentro de \Sexpr{ }.
4. Linguagem de Formatação de Texto Simples
Os três primeiros passos são ajustes simples que podemos aprender e realizar em alguns minutos. A quarta diferença é a mais complexa, mas também a mais poderosa. É o uso da linguagem do Latex fora dos chunks para formatar o nosso texto.
O Latex é mais preciso e controlado do que o Rmarkdown. O padrão para formatar é \comando{texto}, onde o ‘texto’ é impresso no documento, enquanto o ‘comando’ significa o tipo de formatação desejada.
- \textbf{Bold} - Bold
- \textit{italic} - italic
- \underline{underline} - underline
Para gerar listas não ordenadas:
\begin{itemize}
\item Texto 1
\item Texto 2
\end{itemize}
Para gerar listas ordenadas:
\begin{enumerate}
\item Texto 1
\item Texto 2
\end{enumerate}
Títulos e Seções numeradas:
\section{Titulo 1}
\subsection{Subtitulo 1}
\subsubsection{Subsubtitulo 1}
Títulos e Seções não-numeradas:
\section*{Titulo 1}
\subsection*{Subtitulo 1}
\subsubsection*{Subsubtitulo 1}
Equações em Latex
As equações são escritas da mesma forma que em Rmarkdown. Na verdade, o Rmarkdown usa o formato de Latex.
$$\alpha^2 + \beta^2 = \chi^2$$ \[\alpha^2 + \beta^2 = \chi^2\]
$$\frac{\sqrt{1}}{2} * \frac{a}{2b} = \frac{a}{4b}$$ \[\frac{\sqrt{1}}{2} * \frac{a}{2b} = \frac{a}{4b}\] $$\sum_0^{10} x = ...$$ \[\sum_0^{10} x = ...\]
Mais detalhes aqui.
Acentos em Latex
Para inserir acentos, basta abrir um pacote antes de begin{document} e digitar como normal:
\usepackage[latin1]{inputenc}
Página inicial do documento
O nosso tipo de documento ‘article’ nos permite especificar um título e várias outras características antes de \begin{document}.
\title{Relatório}
\author{My Name}
\date{Maio 2019}
Se você compilar o seu PDF agora, não verá nenhuma diferença. Por que? Porque não tem nada diferente entre begin{document} e end{document}. Para fazer estas características aparecem em nosso documento, precisamos inserir o seguinte código depois de \begin{document}:
\begin{document}
\maketitle
Agora, seu resultado deve ser um documento muito profissional.
Tabelas em Knitr
O Latex tem uma gramática de tabelas um pouco chata de preparar. Mas é bem raro recisar digitar ela manualmente. Como em R Markdown, o jeito mais fácil de gerar uma tabela é criar um tibble em R e passar para a função kable(). Após inseri-lo dentro de um chunk no seu documento .Rnw, precisamos fazer apenas mais uma coisa para que ele pereça bonito: adicione o parâmetro results='asis' no header do chunk, como já fizemos com as tabelas de regressão com stargazer no tutorial anterior.
<<results='asis'>>=
library("tidyverse")
library("nycflights13")
flights %>% group_by(origin) %>%
summarize(atraso_media=mean(dep_delay, na.rm=T)) %>%
kable()
@| origin | atraso_media |
|---|---|
| EWR | 15.10795 |
| JFK | 12.11216 |
| LGA | 10.34688 |
Figuras em Knitr
Para inserir gráficos gerados pelo seu código, use a mesma lógica de R Markdown. Crie o gráfico em ggplot dentro de um chunk em seu documento de .Rnw. Não precisamos ajustar nenhum parâmetro do chunk, mas existem várias opções úteis para figuras para controlar o título e tamanho para integrar com Latex.
<<fig.cap="Titulo de Figura", fig.height=4, fig.width=4>>=
flights %>% group_by(origin) %>%
summarize(atraso_media=mean(dep_delay, na.rm=T)) %>%
ggplot() +
geom_col(aes(x=origin, y=atraso_media))
@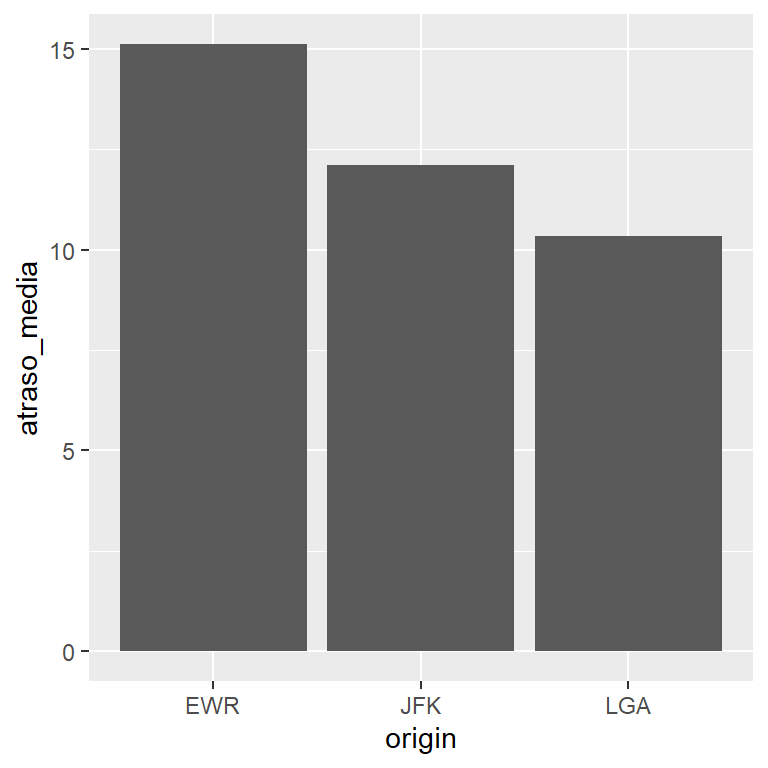
Figure 1: Titulo de Figura
Para aprender mais sobre Latex, pode explorar as guias aqui, aqui e aqui.
Apresentações
O Latex é muito útil para apresentações (slides) profissionais. Há várias opções, mas o mais comum é o estilo ‘beamer’. Comece no início com:
\documentclass{beamer}
Para definir cada ‘slide’ de nossa apresentação, use o seguinte formato:
\begin{frame}
\frametitle{Título do Slide}
Texto, conteúdo normal
Mais texto
\end{frame}
Temos que repetir esta estrutura para cada slide, então 10 slides vão precisar de 10 \begin{frame} e 10 \end{frame}. Se quiser inserir uma pausa, aguardando o usuário para avançar, é só inserir \pause no local apropriado no slide.
Para incluir imagens de um arquivo local salvo na mesma pasta do seu script .Rnw coloque:
\usepackage{graphicx} - antes de \begin{document}
\includegraphics[width=\linewidth]{image.png} - na página/slide onde a imagem deve aparecer
Bibliografias
Um elemento central da pesquisa é a citação das nossas fontes. Uma citação é simplesmente um conjunto de dados sobre um livro/artigo ou outra fonte que colocamos numa nota de rodapé ou apêndice, e um atalho para a citação no texto (frequentemente autor-ano, ex. ‘(Arrow 1961)’).
Felizmente, existe um formato padrão para o armazenamento das citações - num arquivo com extensão ‘.bib’. Várias programas (Zotero, Mendeley, EndNote etc.) podem te ajudar a gerir as citações e gerar arquivos ‘.bib’ que listam todas as citações, então não precisamos nos preocupar com o conteúdo deles. Apenas para referência, eles contém conteúdo tipo:
@article{Arrow1961,
author={Arrow, Kenneth J. and Leonid Hurwicz and Hirofumi Uzawa},
title={Constraint qualifications in maximization problems},
journal={Naval Research Logistics Quarterly},
volume={8},
year=1961,
pages={175-191}
}
O elemento crucial aqui é a primeira entrada depois do primeiro ‘{’: ‘Arrow1961’ - isso é o atalho que vamos usar para citar este artigo para não repetir toda essa informação cada vez que queremos citar o mesmo artigo.
Em R Markdown, o uso de bibliografias é assim:
- Salve o arquivo “.bib” na pasta do seu projeto.
- No cabeçalho, coloque uma linha “bibliography: nome_do_arquivo.bib” para conectar com o arquivo que contém as citações:
---
title: "Exemplo"
output: html_document
bibliography: nome_do_arquivo.bib
---- No lugar apropriado no seu texto simples, quando quiser fazer referência à citação, insira
[@Arrow1961], com o atalho apropriado para a citação desejada. - As referências vão aparecer no final do documento, então pode ser útil inserir um título, como
## Referênciasno final do documento.
Em Knitr (com o Latex) o uso de bibliografias é um pouco mais complexo infelizmente:
- Salve o arquivo “.bib” na pasta do seu projeto.
- Antes de ‘\begin{document}’ coloque o seguinte código:
\usepackage[backend=bibtex, style=authoryear]{biblatex} \addbibresource{Nome_do_Arquivo.bib}
- No lugar desejado do seu texto simples, quando quiser fazer referência à citação, insira
\cite{Arrow1961}, ou\parencite{Arrow1961}se quiser a citação em parênteses, com o atalho apropriado para a citação desejada. - No final do seu documento (imediatamente antes de ‘\end{document}’), coloque
\printbibliography. - Compile o seu script para PDF duas vezes, para que o R consiga incorporar as citações.
Exercício 1: Praticando o Latex
Usando o formato “.Rnw”, crie um PDF com texto simples usando pelo menos cinco das formatações acima (bold, itálico, etc.).
Adicione a famosa equação do teorema de Pitágoras no seu documento.
Adicione uma tabela simples no PDF usando o banco de dados de
weather, resumindo o total de precipitação por mês.
Mostrar Código
weather %>%
group_by(month) %>%
summarize(precip = sum(precip, na.rm = T))
- Adicione um gráfico simples usando o banco de dados
weather, ilustrando temperatura média por aeroporto.
Mostrar Código
weather %>%
group_by(month) %>%
summarize(temp = mean(temp, na.rm = T)) %>%
ggplot() + geom_line(aes(x = month, y = temp, group = 1))
Verifique que o seu documento compila sem erro para PDF.
Ajuste o seu script “.Rnw” acima para gerar uma apresentação do class ‘beamer’ e coloque o texto, a equação, a tabela, e o gráfico em slides diferentes. Compile para PDF de novo.
Controle de Versões com Git
Você já criou um arquivo com nome ‘Final_v23_depois_edits_4b_final_final_v2.doc’? Dois anos depois, você poderia identificar qual versão do documento você enviou para uma colega?? Rastrear mudanças em nossos scripts é desafiador, sobretudo com análises complexas. Felizmente, programadores desenvolveram várias ferramentas para ajudar. Vamos usar ‘Git’ em conjunto com Github, o parceiro online de Git.
O Git/Github é um sistema de controle de versões com três objetivos:
- Disponibilizar um backup online dos seus arquivos;
- Controlar as versões dos scripts e rastrear mudanças;
- Permitir a divulgação/colaboração online com colegas.
Para utilizá-lo (se ainda não executei):
1. Instale git no seu computador.
2. Crie uma conta no site github.
3. Em R, encontre a aba de ‘Terminal’ ao lado de ‘Console’ e ‘R Markdown’. No terminal, digite o seguinte (duas linhas separadas), substituindo os valores de usuário e email com eles que você usou para abrir a conta de Github online:
git config --global user.name 'usuario_de_github'
git config --global user.email 'nome@email.com'
A Lógica de Git
O Git é poderoso e complexo. Neste momento, faz sentido usar apenas as capacidades relevantes para nós. A ideia pode ser separada em dois fluxos de trabalho:
Controle de Versões: Vamos organizar cada mudança substancial em nosso código em um pacote, e indicar para o Git uma breve descrição sobre as mudanças feitas. Ex. “Criando gráfico de pontos dos vôos”. Assim, no futuro, podemos identificar o pacote relevante por meio da sua descrição e ver, linha por linha, quais as mudanças feitas neste pacote. Definir um pacote com o Git se chama um
commit.Backup/Colaboração Online: Vamos enviar todos os pacotes do mesmo projeto para um servidor online (um ‘repositório’) do Github, o qual podemos compartilhar com colegas, ou acessar de um outro computador. Enviar um código ao repositório em Git se chama um
push.
Quando temos mais de uma pessoa trabalhando no mesmo script, o nosso repositório serve como a versão ‘atual’ do script. Isso exige um pouco mais de esforço - temos que baixar a versão atual do repositório antes de trabalhar nele - fazer um pull em Git - e subir as nossas mudanças - com push - quando terminado para disponibilizar para outros.
O gráfico abaixo mostra como funciona o fluxo de código com as várias comandas:
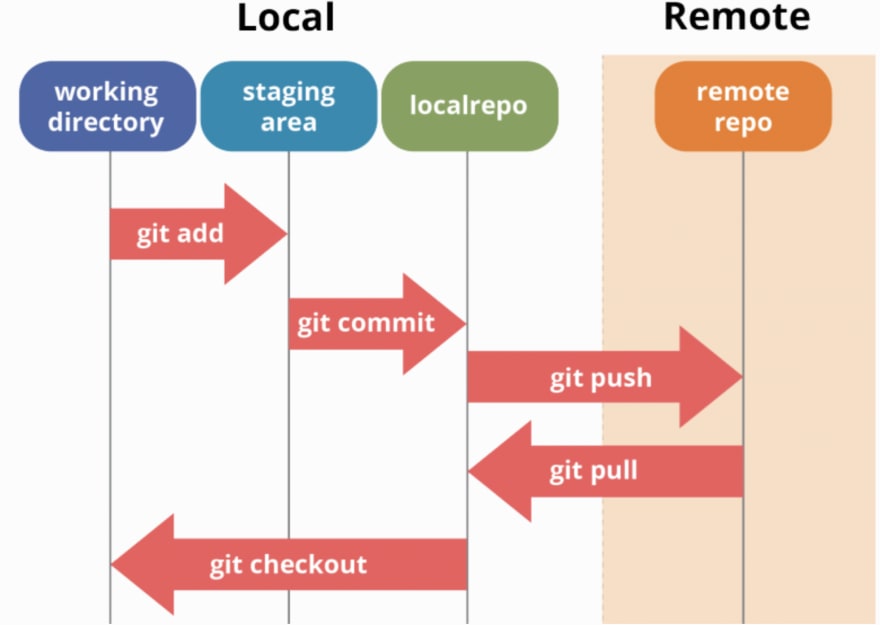
Antes de descrever o que fazer em RStudio, recomendamos adotar o seguinte fluxo de atividades para trabalhar com Git. Cada vez que desejar trabalhar no seu projeto, siga estes passos:
- Abra o projeto em RStudio (o Git funciona apenas dentro de um projeto)
- Execute
Pullda versão mais recente do repositório do Github - Faça as suas mudanças/melhorias no código/relatório em RStudio
- Quando completar uma tarefa discreta, execute
Commitdo pacote de mudanças com uma descrição apropriada - Execute
Pushdo seu pacote/commit para o repositório online - Feche o projeto
Preparando Git/Github em RStudio
Existe um pequeno custo fixo para configurar um projeto para trabalhar com o Git/Github. O primeiro passo é criar um repositório online no Github. Após o login no https://github.com, vá para a aba ‘respositories’, e crie o seu próprio repositório com a opção ‘New’ no site. Dê ao repositório um nome e uma descrição, e finalize sua criação. Verifique se existe um link na página principal do seu repositório, tipo “https://github.com/Usuario/repositorio.git”, o qual você deve copiar. (Se não ver o link, tente clicar na botão verde ‘Code’. Ele deve aparecer em seguida).
Uma alternativa é baixar um repositório de outra pessoa (se você encontrar alguma dificuldade na etapa anterior, por exemplo). Neste caso, você pode usar um repositório do meu github, através do link https://github.com/JonnyPhillips/repositorio_clone.git.
Agora, vamos usar o link para conectar o Github com RStudio. Após abrir o RStudio, vamos ‘clonar’ este repositório online para um projeto local no seu computador: File -> New Project -> Version Control -> Git. Agora, cole o link do repositório de Github como ‘Repository URL’, e escolha o nome da pasta onde o repositório/projeto será baixado no seu computador.
Agora, temos um projeto ligado ao nosso repositório do Github. Na aba ‘files’ do RStudio agora aparece a lista de arquivos no repositório/projeto. Crie um novo script de R Markdown (.Rmd). Neste novo arquivo, coloque o código seguinte para produzir um gráfico super simples. Salve com nome “teste.Rmd” na mesma pasta do seu projeto/repositório (deve ser a pasta padrão).
library("tidyverse")
library("nycflights13")
flights %>% ggplot() +
geom_density(aes(dep_delay)) +
xlim(0, 100)
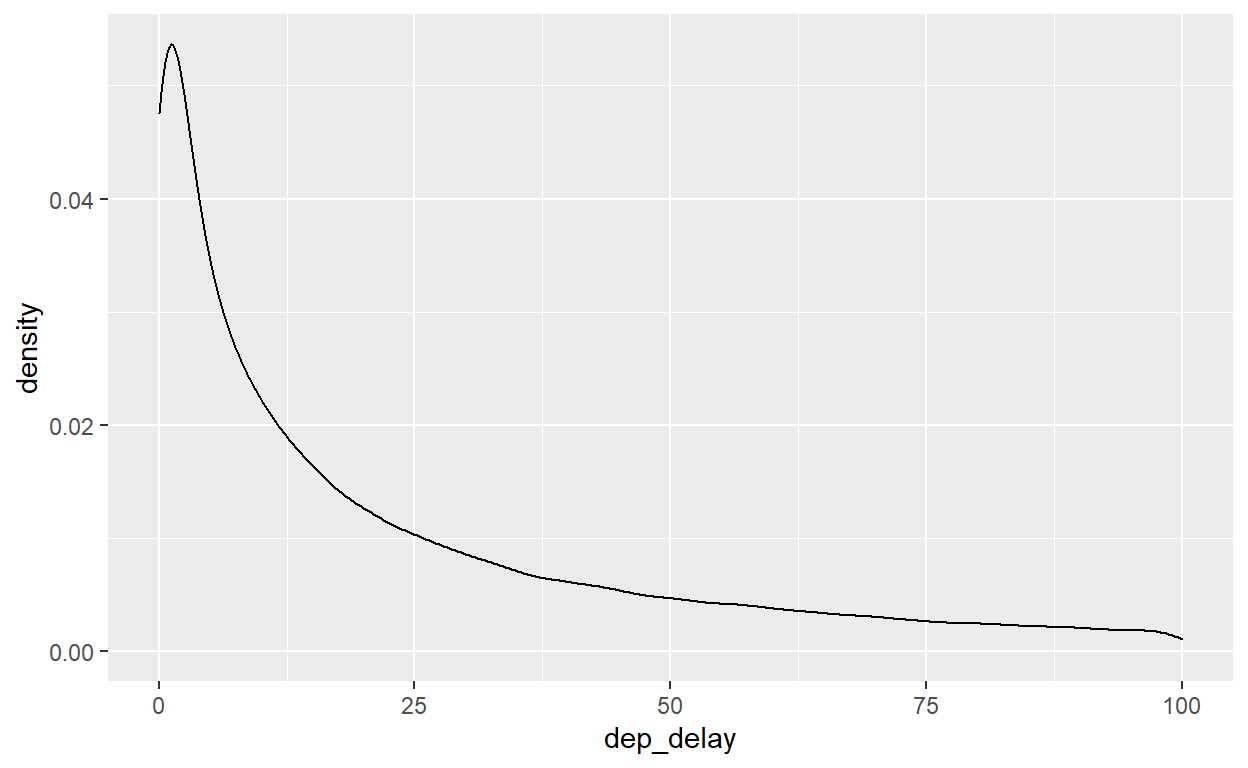
Vá para o repositório na sua conta no github - veja que o arquivo ‘teste.Rmd’ não está presente e só fica no seu computador local. Para sincronizar e ‘atualizar’ o repositório online do github com as nossa mudanças locais, temos que fazer o seguinte:
- Abra a aba ‘Git’ em Rstudio (normalmente na mesma região de ‘Environment’)
- Clique no checkbox ao lado de cada arquivo que você deseja atualizar, incluindo ‘teste.Rmd’. O ‘Status’ vai mudar para “A” (que significa ‘Adicionado’), ou caso você esteja atualizando um arquivo já existente, “M” (que significa ‘Modificado’). Cada vez que você ajusta e salva um arquivo no projeto local, o arquivo vai aparecer na lista nessa aba de Git.
- Clique em ‘Commit’ na aba do Git e veja que abriu uma caixa que mostra todas as diferenças entre o nosso código local e o código do arquivo no github online: aditivos em verde e remoções em vermelho. A única coisa que temos que fazer nessa caixa é adicionar uma mensagem que descreve o conteúdo da mudança feita, ex. “Adicionar gráfico de atrasos”, e clicar em
Commit. Quando terminado, feche as caixas. A aba do Git deve mostrar a notícia de “Your branch is ahead of ‘origin/master’ by 1 commit”. Isso significa que o seu novo script foi cadastrado no fluxo de trabalho do projeto (o controle de versão), mas ainda não foi sincronizado online. - O passo final é clicar em
Pushna aba ‘Git’ em RStudio (a seta verde). Isso sincroniza as mudanças contidas nocommitcom a versão dos arquivos online, disponibilizando elas para os seus colegas.
Agora vá para o repositório na sua conta do Github online, atualize a página, e confira que o novo arquivo ‘teste.Rmd’ aparece. (É possível que você precisa aguardar alguns minutos para ele aparecer, mas normalmente é imediato). Se você entrar no link de ‘1 commit’ na linha azul na página do seu repositório, poderá ver todos os seus commits anteriores, e os detalhes individuais de mudanças de código em cada um. Também é possível clicar em cada arquivo e depois em ‘History’ (histórico) rastrear todas as mudanças anteriores.
Parabéns, agora o seu trabalho está seguro com um backup online, documentado com o histórico de versões rastreável, e acessível para todos os seus colegas!
Habilidade Básica de Programação: Privacidade
O padrão para Github é que os repositórios fiquem abertos para todo o mundo acessar, encorajando transparência e reprodutibilidade. Mas tome cuidado para verificar que o seu projeto não contém dados confidenciais ou alugma outra informação pessoal.
Se você tiver uma conta paga, ou uma conta estudantil, pode tornar a sua conta privada e adicionar apenas os colaboradores desejados - em Github vá para Settings -> Make Private.
Exercício 2: Usando Git
Crie um novo repositório na sua conta de Github e conecte (‘clonar’) com um novo projeto no seu RStudio.
Copie o seu script de Exercício 1 acima (o .Rnw) para a pasta local do seu projeto novo criado no passo anterior.
Adicione mais um gráfico ao seu script, mostrando a umidade média por mês do banco de dados de
weather.
Mostrar Código
weather %>%
group_by(month) %>%
summarize(humid = mean(humid, na.rm = T)) %>%
ggplot() + geom_line(aes(x = month, y = humid, group = 1))
Usando a aba de Git, execute
Commitcom a versão atualizada do script Rnw, atribuindo uma descrição apropriada.Execute
Pushdas mudanças para o seu repositório do Github online. Verifique que o novo arquivo está atualizado no repositório da sua conta deo Github.
Bibliotecas Reproduzíveis: Renv
A última ferramenta listada aqui é menos usada, mas atende um problema que já é familiar para vocês - complexidades e conflitos de bibliotecas e funções de R. Essa seção é optativa porque não é essencial para o seu trabalho pessoal, mas pode ser útil para projetos maiores e colaborativos.
O que vai acontecer se mandamos o nosso script de R Markdown para uma colega? Vai funcionar? Será reprodutível? Frequentemente não. Mesmo que eles tenham exatamente os mesmos arquivos e usem a mesma versão de R e RStudio, não podemos garantir que eles terão acesso às mesmas funções que usamos para fazer nosso script. Lembre-se que tivemos que instalar cada uma das nossas bibliotecas (install.packages()) mas essas linhas de instalação não ficam em nosso script - se o receptor não tiver feito o mesmo ou tiver uma versão diferente de uma biblioteca (eles estão atualizadas frequentemente), nosso código vai quebrar.
Para resolver isso, podemos usar o pacote renv para garantir reprodutibilidade de bibliotecas. O renv salva as bibliotecas na pasta do seu projeto, e documenta as versões que estão sendo usadas. No modo interativo, rodamos a função init() uma vez quando começamos um novo projeto. Em seguida, quando você desejar gravar o status das suas bibliotecas (normalmente depois de abrir novas bibliotecas com library()), você pode usar snapshot().
Se você mander o seu script para uma outra pessoa, ou outra máquina, para recuperar exatamente as bibliotecas e versões que você tinha instalada, é só rodar restore() na nova maquina e tudo será instalado de forma idêntica.
Muita coisa, não é mesmo? Mas, quando combinados, o R, o Latex, o Git e o Renv permitem relatórios reproduzíveis que garantem o histórico e a integridade do seu relatório ou artigo profissional.
Leitura para Tutorial 11
Antes da próxima aula, por favor leia R 4 Data Science, Capítulos 20 e 21 sobre Vetores e Iteração